Pesquisa registra biodiversidade microbiana utilizando espectrometria de massas em comunidades de fungos e bactérias associadas a plantas.
Por Edson Rodrigues Filho*
Uma das mais importantes habilidades dos micro-organismos é a capacidade de adaptação aos substratos onde eles se associam para obter os nutrientes que formam a base do seu desenvolvimento e manutenção dos seus ciclos de vida. O uso de um sistema enzimático quase sempre muito complexo permite-lhes sobrepor barreiras impostas por seus hospedeiros e se instalar em outras formas de vida ou ocupar posições em diversos nichos ecológicos. Essa adaptação é quase sempre envolvida de uma extensa batalha química consumindo ou produzindo um grande arsenal de substâncias que se acumulam na natureza. Essas substâncias podem causar grandes danos direta ou indiretamente ao homem (micotoxinas carcinogênicas, fitotoxinas, etc), ou o ajudar a curar doenças causadas por outros micro-organismos (antibióticos, anticancer, etc). Micro-organismos podem beneficiar o homem indiretamente, contribuindo para a produtividade agrícola quando eles enriquecem o solo e auxiliam o desenvolvimento de plantas importantes para o consumo humano.
Embora os micro-organismos estejam presentes na maioria das formas de vida no planeta, apenas uma pequeníssima parcela deles é conhecida, e somente uma parcela ainda menor é dominada e manipulada pelo homem. Ainda é ínfimo o conhecimento e controle de consequências negativas das influências de micro-organismos na vida humana, bem como ainda é desprezível o número de explorações biotecnológicas diante da vasta diversidade microbiana no planeta.
Um dos problemas mais importantes que todos os pesquisadores brasileiros envolvidos nessas pesquisas enfretamos neste momento reside nas dificuldades para identificação de micro-organismos, o que tem resultado em falhas nos direcionamentos dos estudos posteriores às coletas; falta de um catálogo de referência para cepas coletadas, re-estudo de uma mesma cepa microbiana por diferentes grupos, etc. Uma única folha de uma planta, ou alguns poucos miligramas de solo, podem conter um número muito grande de micro-organismos. Escolher qual deles estudar tem sido uma tarefa difícil e muitas vezes materiais biológicos importantes são descartados e desperdiçados. Embora em cada estudo seja isolada uma vasta gama de micro-organismos, apenas uma pequena porcentagem deles tem sido identificada e reportada. A identificação de micro-organismos deve preceder qualquer estudo de exploração biotecnológica, seja para orientar para os melhores potenciais de aplicação, ou para evitar sobreposição de estudos por diferentes grupos.
Identificações de micro-organismos sempre foram tarefas difíceis. Inicialmente isso foi exclusivamente feito pela inspeção macro e micro morfológica (Figura 2), o que depende imperativamente de grande especialização dos profissionais envolvidos nessa tarefa. Depois vieram as técnicas de biologia molecular como uma das mais poderosas ferramentas para uma identificação segura no nível genômico. Essas duas estratégias, ou combinações delas, são sempre muito demoradas e às vezes inconclusivas.
Impressões digitais dos micro-organismos
Recentemente, uma nova tecnologia para identificar micro-organismos emergiu com um grande destaque devido à agilidade e eficiência. Essa nova tecnologia usa principalmente a análise de algumas moléculas boas caracterizadoras como, por exemplo, algumas proteínas ribossomais, através de espectrometria de massas com ionização por lazer (MALDI/TOF – MS). Nessa abordagem (Figura 3), células microbianas são depositadas em uma placa de amostragem e juntada com uma substância fotoativa (matriz); a incidência de raios luminosos (laser) produz íons a partir das biomoleculas presentes nesse material biológico e esses íons são analisados de acordo com sua massa e carga, gerando milhares de espectros que são somados formando um “espectro principal” (MSP – main spectrum) para cada amostra; esses dados são avaliados estatisticamente usando programas computacionais. Os MSPs são usualmente tratados como uma impressão digital do micro-organismo. Essa tecnologia já é bastante usada em microbiologia médica, onde tem sido importantíssima para identificação de infecções bacterianas hospitalares, e está sendo agora adaptada para a identificação de fungos. Depois de cumpridas as etapas de certificação de cepas e aumento da base com dados confiáveis, esse sistema pode representar uma ferramenta de extrema utilidade para catalogar e identificar milhares de micro-organismos, e com isso racionalizar os estudos que exploram biodiversidade microbiana, bem como para os próprios estudos proteômicos e genéticos de micro-organismos feitos pelos taxonomistas.

As biomoléculas que são amostradas em análises como essas, em muitos casos são assumidas como pequenas proteínas. As proteínas produzidas no ribossomo têm quase sempre essa faixa de massas. Além disso, essas moléculas são codificadas em uma região bastante conservada do genoma, sendo então de muito boa utilidade taxonômica. Em bactérias, a quantidade dessas proteínas é sempre muito grande, podendo chegar a mais de 20% da biomassa seca. Por isso, os dados de MALDI/TOF de células bacterianas são sempre muito bons e fiéis às espécies. A identificação de bactérias por essa técnica de espectrometria de massas já vem sendo aprovada para ambientes hospitalares em diversos países, já com bons indicativos de aprovação o Brasil.
Por outro lado, para fungos a diversidade de estruturas morfológicas e de modos reprodutivos resulta em um grande desafio para se obter bons perfis espectrais que possam ser correlacionados com as espécies. Bons MSPs são gerados para fungos que produzem esporos em abundância. No entanto, ainda assim para alguns gêneros podemos obter excelentes correlações espectrais mesmo com mudanças de meios de cultura para o crescimento das células (Figura 4).
Dados como estes mostrados na Figura 4 podem ser usados para estudos de filogenia, comparando espécies e linhagens de um mesmo gênero de fungos (como fizemos para o gênero Clonostachyes [1] recentemente, ou ainda para diferentes gêneros (Figura 5). Enfim, essa estratégia pode ser usada com sucesso para diferenciação de espécies microbianas. Algumas análises filogenéticas por MALDI/TOF podem correlacionar muito bem com aquelas obtidas por biologia molecular para os mesmos micro-organismos. No entanto, como as características avaliadas são diferentes, algumas discrepâncias são admitidas.
Estratégias de indentificação rápida
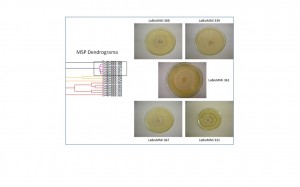
Fungos ainda não identificados podem ser comparados entre si, permitindo uma avaliação da seletividade e/ou especificidade da colonização de diversos hospedeiros. As vantagens para isso são muitas. Principalmente, a obtenção dos dados não requer esforços de profissionais com grande experiência em microbiologia, taxonomia, etc. Além disso, a obtenção dos dados e suas análises são simples e rápidas. Pequenas variações morfológicas que possam levar à suspeita de se tratar de espécies diferentes são também refletidas nas comparações dos MSPs (Figura 6). Dessa forma, é possível comparar todos os micro-organismos isolados de uma ou várias plantas, e re-agrupá-los conforme gêneros/espécies. Quando cepas autênticas (identificadas por outros métodos) estão depositadas no banco do espectrômetro, é possível a identificação de cada espécie por confronto direto. Caso contrário, as análises por MALDI/TOF serão úteis somente para a indicação de que cepas entre as várias coletadas devem ser enviadas para identificação por sequenciamento genômico. Isso poupa muito tempo dos profissionais envolvidos e recursos.


No nosso projeto, estamos envolvidos com a aquisição de dados do perfil de biomoléculas por MALDI/TOF de fungos e bactérias coletados e diversos ambientes. Embora o Brasil seja um país em que está documentada uma das maiores biodiversidade de plantas e pequenos animais, insetos, etc, muito pouco ainda é conhecido sobre a diversidade de micro-organismos vivendo em associação com os diversos substratos, vivos ou não, encontrados no nosso território. Os pesquisadores do Laboratório de Bioquímica Micromolecular de Micro-organismos (LaBioMMi), instalado no Depto. de Química da Universidade Federal de São Carlos (UFSCar) desde 1996, vêm coordenando uma rede de estudos para a documentação da biodiversidade microbiana brasileira. O histórico de trabalho do grupo tem certa ênfase nos micro-organismos endofíticos, mas o estudo dos vários aspectos do metabolismo microbiano vem sendo estendido também para outros tipos de associações. Esses estudos fazem parte do programa Sisbiota-Brasil. As atividades nessa rede se referem ao estabelecimento de estratégias de identificação rápida, através do perfil proteômico usando espectrometria de massas por MALDI/TOF, padronização de metodologias de coleta, identificação e conservação de micro-organismos, bem como indicações de possíveis aplicações biotecnológicas. Fazem parte da rede pesquisadores de onze instituições de ensino superiores (IES) brasileiras, incluindo duas universidades de Manaus, uma de Belém, e uma de Fortaleza. O projeto tem sido financiado pelo CNPq e pela CAPES¹.
Alguns dos resultados já divulgados até aqui envolvem os estudos do microbioma e seu metaboloma de frutas comestíveis, como goiaba (Figura 7) [2]; coleções de micro-organismos produtores de antibióticos [3]; micro-organismos coletados nas plantas medicinais Syzygium cumini [4] e Alternanthera brasiliana [5]; e também investigações de bactérias coletadas em ambientes hospitalares [6].
Entre as demais plantas que vêm sendo amostradas mais recentemente, estão Rosmarinus officinalis (Alecrim), Piper aduncum, Cinnamomon spp., Podocarpos spp., Cupressus lusitanica e Sapinduas saponaria, coletadas na região de São Carlos – SP, além das plantas tradicionalmente estudadas no grupo LaBioMMi como Melia azedarach e Murraya paniculata; várias espécies coletadas na região de Manaus; regiões de minas no estado do Pará; sedimentos marinhos na costa do Ceará; e insetos da região de Londrina – Paraná
Conforme nossas expectativas, a quantidade e variedade de micro-organismos que convivem com as plantas são sempre surpreendentes. Em vários casos estudados até aqui, há grande variedade de espécies microbianas associadas à a maioria das plantas. Mas há também espécies que são colonizadas por um único ou apenas poucos micro-organismos, como Sapindus saponaria. Os gêneros Xylaria, Alternaria, Pestalotiopsis, Penicillium, Aspergillus, Mucor e Rhizopus estão entre os fungos filamentosos mais frequentemente encontrados nos estudos
*Coordenador do projeto: “Rede de estudos em biodiversidade microbiana: isolamento, desenvolvimento de estratégias de identificação rápida, conservação, interações entre espécies, e explorações biotecnológicas” e membro do Laboratório de Bioquímica Micromolecular de Micro-organismos – LaBioMMi, do Departamento de Química, da Universidade Federal de São Carlos.
¹Universidade Federal do Pará (UFPA); Universidade Federal do Amazonas (UFAM); Universidade Estadual do Amazonas (UEA); Universidade Federal do Ceará (UFC); Universidade Federal de Minas Gerais (UFMG); Universidade Federal de Lavras (UFLA); Universidade Tecnológica Federal do Paraná (UTFPR). Universidade Estadual Norte Fluminense (UENF) Universidade Estadual Paulista (UNESP) Instituto Federal de Sergipe (IFS)
